Molecular Modelling
Molecular Modelling von großen Membran-Protein-Komplexen
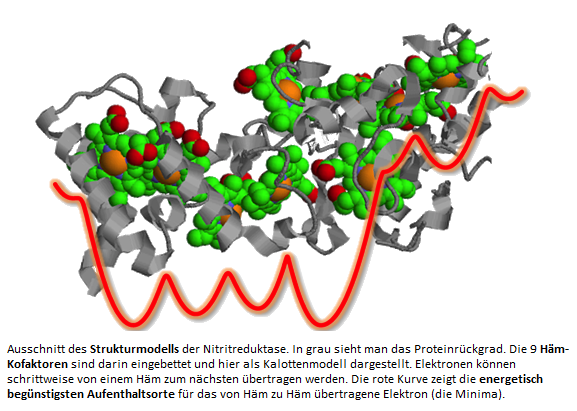 WissenschaftlerInnen der Forschungs-gruppe von Thorsten Koslowski haben den Mechanismus des Elektronentransfers in der Nitritreduktase mit Hilfe von Computer-simulationen beleuchtet. Dieser Membran-Protein-Komplex überträgt in Pflanzen und Bakterien Elektronen über eine Kette von Häm-Kofaktoren auf ein Nitrit-Anion. So katalysieren diese Enzyme die Reduktion zum Ammonium-Ion, die biologisch wichtigste Stickstoffquelle für das Wachstum der Pflanzen. Die WissenschaftlerInnen konnten bestimmen, auf welchen in einer Reihe liegenden Häm-Gruppen der Aufenthalt für das Elektron energetisch am günstigsten ist. An diesen Hämen gebunden werden die Elektronen für kurze Zeit gespeichert. Am Ende des Transportweges (im Bild auf der rechten Seite) verhindert eine Energie-barriere, dass die Elektronen weiterwandern und verloren gehen. Erst wenn ein Nitrit-Anion im aktiven Zentrum bindet, fließen die Elektronen weiter und reduzieren das Nitrit-Anion zum Ammonium-Ion.
WissenschaftlerInnen der Forschungs-gruppe von Thorsten Koslowski haben den Mechanismus des Elektronentransfers in der Nitritreduktase mit Hilfe von Computer-simulationen beleuchtet. Dieser Membran-Protein-Komplex überträgt in Pflanzen und Bakterien Elektronen über eine Kette von Häm-Kofaktoren auf ein Nitrit-Anion. So katalysieren diese Enzyme die Reduktion zum Ammonium-Ion, die biologisch wichtigste Stickstoffquelle für das Wachstum der Pflanzen. Die WissenschaftlerInnen konnten bestimmen, auf welchen in einer Reihe liegenden Häm-Gruppen der Aufenthalt für das Elektron energetisch am günstigsten ist. An diesen Hämen gebunden werden die Elektronen für kurze Zeit gespeichert. Am Ende des Transportweges (im Bild auf der rechten Seite) verhindert eine Energie-barriere, dass die Elektronen weiterwandern und verloren gehen. Erst wenn ein Nitrit-Anion im aktiven Zentrum bindet, fließen die Elektronen weiter und reduzieren das Nitrit-Anion zum Ammonium-Ion.
Durch Molecular Modelling kann die Wissenschaft sehr schnelle chemische Reaktionen, wie den Elektronentransfer in Proteinen, im Nanosekundenbereich untersuchen. Grundlage für die Simulationen sind die experimentell ermittelten Strukturinformationen der Proteine auf atomarer Ebene, die auch für große Membran-Protein-Komplexe erhalten werden können. Der Proteinkomplex kann als Ganzes simuliert werden. Dies erfordert aber bei so großen Proteinkomplexen wie die Nitritreduktase eine sehr hohe Rechenleistung. Ein anderer Ansatz ist, das System auf die für die Untersuchung relevanten Einheiten zu reduzieren, z.B. auf eine einzelne Proteinkette mit Kofaktoren. Die hier untersuchte Nitritreduktase aus dem Bakterium D. vulgaris besteht aus 6 Proteinketten. Jeweils 4 und 2 dieser Ketten haben die gleiche Struktur. Die Simulationen der WissenschaftlerInnen zeigen, dass die kleinste funktionelle Einheit des Enzyms ein Heterodimer aus zwei unterschiedlichen Proteinketten mit insgesamt 9 Häm-Kofaktoren ist (siehe Abbildung). Die Größe des Enzyms mit 6 Proteinketten und 28 Hämen ermöglicht eine Zwischenspeicherung der für die Reduktion von Nitrit benötigten Elektronen.
Originalveröffentlichung:
Sehee Na, Anna Bauß, Michael Langenmaier und Thorsten Koslowski
Thermodynamic integration network study of electron transfer: from proteins to aggregates.
Physical Chemistry Chemical Physics 19 (2017), 18938-18947
Diese Veröffentlichung wurde vom zuständigen Herausgeber und den Gutachtern der Fachzeitschrift „ Physikal Chemistry Chemical Physics“ zu den „Hot Articles 2017“ gewählt!
Kontakt:
Prof. Dr. Thorsten Koslowski
Theoretische Chemie
Institut für Physikalische Chemie
Albert-Ludwigs-Universität Freiburg
Tel.: (+49) 761 - 203 - 6182
E-Mail: thorsten.koslowski@physchem.uni-freiburg.de

